QTM 347 HW2 (2599822, 2539356, 2453794, 2396043)
March 19, 2024
[ ]: import numpy as np
import matplotlib.pyplot as plt
from ISLP import load_data
import warnings
warnings .filterwarnings( 'ignore ')
0.1 Q1.
a) The probability of not selecting the first observation is 1-1/n
b) The probability of not selecting the first observation is 1-1/n. Since the order doesn’t matter in bootstrap, every draw has same probability distribution
c) (1-1/10)^10
d) (1-1/100)^100
e) (1-1/1000)^1000
f)
[ ]: n_values_specific = np .array([10, 100, 1000])
probabilities_specific = (1 - 1/n_values_specific) ** n_values_specific
n_values_range = np .arange(1, 100001)
probabilities_range = (1 - 1/n_values_range) ** n_values_range
plt .figure(figsize=(10, 6))
plt .plot(n_values_range, probabilities_range, label= 'Probability not in sample ')
plt .xscale( 'log ')
plt .xlabel( 'n (log scale) ')
plt .ylabel( 'Probability ')
plt .title( 'Probability that the first observation is not in the bootstrap␣ ↪ sample ')
plt .grid(True, which="both", ls="--")
plt .show()
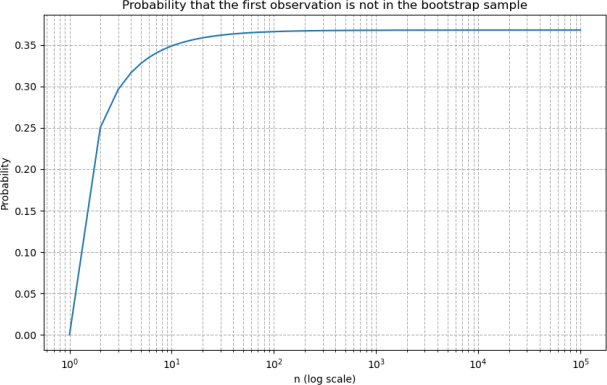
We can see a pattern of convergence. This pattern illustrates an important concept in bootstrap sampling: as the size of the original sample increases, the probability of any specific observation being omitted from a bootstrap sample approaches a limit. This reflects the balance between the increasing number of opportunities for an observation to be included in the bootstrap sample and the constant probability of it being selected in any single draw.
0.2 Problem 2:
[ ]: college = load_data("College")
college["Accept.Rate"] = college["Accept"]/college["Apps"]
college = college .drop("Accept", axis=1)
college = college .drop("Apps", axis=1)
[ ]: ## a)
mu_hat = college["Accept.Rate"] .mean()
mu_hat
[ ]: 0.7469277072775414
[ ]: ## b)
standard_error= college["Accept.Rate"] .std()/np .sqrt(len(college["Accept .
↪Rate"]))
print("The estimated standard error is: ",standard_error)
The estimated standard error is: 0.005277323728707518
[ ]: ## c)
def boot_mean(X, B=1000, seed=0):
rng = np .random .default_rng(seed)
first_, second_ = 0, 0
n = X .shape[0]
print("mean", np .mean(X))
for _ in range(B):
idx = rng .choice(n, n, replace=True)
value = np .mean(X .loc[idx])
first_ += value
second_ += value**2
return np .sqrt(second_ / B - (first_ / B)**2)
se_mu_hat_bootstrap=boot_mean(college["Accept.Rate"])
mean 0.7469277072775414
[ ]: ## d)
from scipy.stats import ttest_1samp
from scipy import stats
import statsmodels.api as sm
[ ]: confidence_interval = [mu_hat - 2*se_mu_hat_bootstrap, mu_hat +␣ ↪2*se_mu_hat_bootstrap]
confidence_interval
[ ]: [0.7364686641045017, 0.7573867504505811]
[ ]: t_statistic, p_value = ttest_1samp(college[ 'Accept.Rate '], mu_hat)
se = np .std(college["Accept.Rate"]) / np .sqrt(len(college["Accept.Rate"]))
df = len(college["Accept.Rate"]) - 1
t_critical = stats .t .ppf(0.975, df)
confidence_interval_ttest = (mu_hat - t_critical * se, mu_hat + t_critical * se)
print("confidence interval from ttest is␣
↪ ",confidence_interval_ttest,"\nconfidence indeterval from bootstrap␣ ↪ is",confidence_interval)
confidence interval from ttest is (0.7365748535300279, 0.7572805610250549)
confidence indeterval from bootstrap is [0.7364686641045017, 0.7573867504505811]
The confidence intervals from the one-sample t-test and the bootstrap method are very close, indicating consistent estimates for the mean acceptance rate. This similarity suggests that the bootstrap method is a robust alternative to the traditional t-test approach. Differences between the intervals can be attributed to the variability in bootstrap resampling and the approximation methods used. The bootstrap method is less reliant on distributional assumptions, making it versatile for various data types. Both methods provide reliable evidence about the mean acceptance rate, demonstrating the effectiveness of these statistical tools in analysis.
[ ]: ## e)
def boot_OLS(X, Y, B=1000, seed=0):
rng = np .random .default_rng(seed)
n = X .shape[0]
first_, second_ = np .zeros((2,)), np .zeros((2,))
for _ in range(B):
idx = rng .choice(n, n, replace=True)
Y_ = Y .loc[idx]
X_ = X .loc[idx]
# add constant
X_ = sm .add_constant(X_)
# sm.OLS does not automatically include an intercept term
value = sm .OLS(Y_, X_) .fit() .params .values
first_ += value
second_ += value * value
return np .sqrt(second_ / B - (first_ / B)**2)
se_beta = boot_OLS(college["Top10perc"], college["Accept.Rate"])
print(f"Bootstrap standard errors: Intercept ( 0): {se_beta[0]}, Coefficient␣ ↪ ( 1): {se_beta[1]}")
Bootstrap standard errors: Intercept ( 0): 0.00934089871787182, Coefficient
( 1): 0.0003245674075839749
[ ]: ## f)
X = sm .add_constant(college["Top10perc"])
Y = college["Accept.Rate"]
model = sm .OLS(Y, X) .fit()
se_beta 0 ols = model .bse[0]
se_beta 1 ols = model .bse[1]
print(f"Standard error of intercept ( 0): {se_beta 0 ols}")
print(f"Standard error of coefficient ( 1): {se_beta 1 ols}")
Standard error of intercept ( 0): 0.00860399673826018
Standard error of coefficient ( 1): 0.00026300038151207936
The bootstrap standard errors are slightly higher than those calculated using the standard formula in sm.OLS(). This discrepancy can occur due to the bootstrap method’s nature, which involves resampling with replacement to create many “pseudo-datasets.” These pseudo-datasets capture the variability in the data, potentially leading to slightly higher estimated standard errors if the original dataset has certain peculiarities or is not perfectly representative of the population.
[ ]: ## g)
from sklearn.neighbors import KNeighborsRegressor
[ ]: def bootstrap_knn_se(X, y, predict_point, n_bootstrap=1000, k_neighbors=10,␣ ↪ seed=0):
rng = np .random .default_rng(seed)
predictions = []
# Ensure X and y are numpy arrays
X = X .values
y = y .values
for _ in range(n_bootstrap):
# Bootstrap sampling
indices = rng .choice(len(X), len(X), replace=True)
X_sample, y_sample = X[indices], y[indices]
# Reshape X_sample for scikit-learn compatibility
X_sample_reshaped = X_sample .reshape(-1, 1)
# KNN regression
knn = KNeighborsRegressor(n_neighbors=k_neighbors)
knn .fit(X_sample_reshaped, y_sample)
# Predicting for Top10perc=76
pred = knn .predict(np.array([[predict_point]]))
predictions .append(pred[0])
# Compute standard error (standard deviation of bootstrap predictions)
return np .std(predictions, ddof=1)
se_pred_accept_rate = bootstrap_knn_se(college["Top10perc"], college["Accept .
↪Rate"], 76, n_bootstrap=1000, k_neighbors=10, seed=42)
se_pred_accept_rate
[ ]: 0.03400926935401351
[ ]: se_pred_accept_rate_5 = bootstrap_knn_se(college["Top10perc"], college["Accept .
↪Rate"], 76, n_bootstrap=1000, k_neighbors=5, seed=42)
se_pred_accept_rate_50 = bootstrap_knn_se(college["Top10perc"], college["Accept .
↪Rate"], 76, n_bootstrap=1000, k_neighbors=50, seed=42)
print("K= 5 provide a SE of ",se_pred_accept_rate_5,"\nK=50 provide a SE␣ ↪of",se_pred_accept_rate_50)
K= 5 provide a SE of 0.038934844284861866
K=50 provide a SE of 0.02894815513257801
Lower K (K=5) results in a higher standard error. This suggests that with fewer groups (or resamples), there’s more variability in the estimate. This could be because each group has to cover a wider range of the data, leading to less precise estimates.
Intermediate K (K=10) shows a reduction in standard error compared to K=5, indicating that increasing the number of groups (or resamples) can lead to more stable and reliable estimates, as the variability due to random sampling is reduced.
Higher K (K=50) further reduces the standard error to the lowest level among the three scenarios. This indicates that as K increases, the precision of the estimate improves, likely due to the increased granularity in sampling or cross-validation. Each subset of the data is smaller, leading to potentially more accurate and less variable estimates of the model’s performance or the parameter being estimated.
0.3 Problem 3
[ ]: college[ 'Private_numeric '] = college[ 'Private '] .map({ 'Yes ' : 1, 'No ' : 0})
[ ]: import pandas as pd
import statsmodels.api as sm
# Assuming 'college' is your DataFrame and it's already loaded
response = college[ 'Accept.Rate ']
predictors = college .columns .drop([ 'Accept.Rate ', 'Private ']) # Exclude the␣
↪ response and original Private column
best_r2_a = -float("inf")
best_model_a = None
# (a) Best Subset Selection for One-Predictor Model
for predictor in predictors:
X = sm .add_constant(college[[predictor]]) # Ensure X is a DataFrame
model = sm .OLS(response, X) .fit()
if model .rsquared > best_r2_a:
best_r2_a = model .rsquared
best_model_a = (predictor, model)
print(f"Best one-predictor model is based on {best_model_a[0]} with R^2 =␣ ↪{best_r2_a}")
Best one-predictor model is based on Top10perc with R^2 = 0.22913012842545943
0.3.1 (b)
[ ]: from itertools import combinations
best_r2_b = -float("inf")
best_model_b = None
best_predictors_b = None
# Generate all combinations of two predictors
for predictor_pair in combinations(predictors, 2):
X = sm .add_constant(college[list(predictor_pair)]) # Make sure to convert␣ ↪ the tuple to a list
model = sm .OLS(response, X) .fit()
if model .rsquared > best_r2_b:
best_r2_b = model .rsquared
best_model_b = model
best_predictors_b = predictor_pair
print(f"Best two-predictor model is based on {best_predictors_b} with R^2 =␣ ↪{best_r2_b}")
Best two-predictor model is based on ('Top10perc', 'Private_numeric') with R^2 = 0.2566214603917484
0.3.2 (c)
[ ]: best_predictor_a = best_model_a[0] # Assuming this is the best predictor from␣ ↪part (a)
best_r2_c = best_r2_a # Start with the R^2 from the best one-predictor model best_model_c = best_model_a[1] # And the model itself
best_predictors_c = best_predictor_a # Start with the best single predictor
# Iterate over the remaining predictors to find the best two-predictor model
for predictor in predictors:
if predictor != best_predictor_a: # Skip the predictor used in the best␣
↪ one-predictor model
X = sm .add_constant(college[[best_predictor_a, predictor]])
model = sm .OLS(response, X) .fit()
if model .rsquared > best_r2_c:
best_r2_c = model .rsquared
best_model_c = model
best_predictors_c = (best_predictor_a, predictor)
print(f"Best two-predictor model (forward stepwise) is based on␣ ↪{best_predictors_c} with R^2 = {best_r2_c}")
Best two-predictor model (forward stepwise) is based on ('Top10perc',
'Private_numeric') with R^2 = 0.2566214603917484
0.3.3 d
In part (a), I fit 15 models. In part (b), I fit 105 models considering all pairs of predictors. In part (c), using forward stepwise selection, at least 29 models are fitted in the process to find the best two-predictor model.
The fact that the best two-predictor model is the same regardless of the selection method (exhaus- tive search vs. forward stepwise) indicates that “Top10perc” and “Private__numeric” are strong predictors when used together compared to other combinations or individual predictors. The in- crease in R2R^2R2 from the best one-predictor to the two-predictor model shows that adding “Private__numeric” provides a significant incremental predictive value beyond what “Top10perc” offers alone.
0.4 Problem 4
0.4.1 a)
[ ]: from sklearn.model_selection import train_test_split, KFold
[ ]: X = college .drop([ 'Accept.Rate ', 'Private '], axis=1)
y = college[ 'Accept.Rate ']
# Splitting the dataset into training (80%) and test (20%) sets
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2,␣ ↪random_state=50)
0.4.2 b)
[ ]: from sklearn.linear_model import LinearRegression
from sklearn.metrics import mean_squared_error
# Fitting the linear regression model
linear_model = LinearRegression()
linear_model .fit(X_train, y_train)
# Making predictions on the test set
y_pred_linear = linear_model .predict(X_test)
# Calculating and reporting the test MSE
linear_test_mse = mean_squared_error(y_test, y_pred_linear)
print(f"Linear Regression Test MSE: {linear_test_mse}")
Linear Regression Test MSE: 0.019270389442537825
0.4.3 c)
[ ]: from sklearn.linear_model import ElasticNetCV
from sklearn.preprocessing import StandardScaler
from sklearn.pipeline import Pipeline
from sklearn.metrics import mean_squared_error
import matplotlib.pyplot as plt
[ ]: import sklearn.linear_model as skl
lambdas = 10**np .linspace(8, -2, 100) / y_train .std()
college[ 'Accept.Rate ']
kfold = KFold(n_splits=5, shuffle=True, random_state=50)
ridgeCV = ElasticNetCV(alphas=lambdas, l1_ratio=0, cv=kfold)
scaler = StandardScaler()
pipeCV = Pipeline(steps= [( 'scaler ', scaler), ( 'ridge ', ridgeCV)])
pipeCV .fit(X_train, y_train)
y_pred = pipeCV .predict(X_test)
test_mse = mean_squared_error(y_test, y_pred)
print(f"Test MSE: {test_mse}")
tuned_ridge = pipeCV .named_steps[ 'ridge ']
best_alpha = tuned_ridge .alpha_
print(f"Best alpha ( ) selected by cross-validation: {best_alpha}")
Test MSE: 0.019265604345980403
Best alpha ( ) selected by cross-validation: 0.08932201930628549
0.4.4 d)
[ ]: lambdas = 10**np .linspace(8, -2, 100) / y_train .std()
K = 5
kfold = KFold(K, random_state=50, shuffle=True)
lassoCV = ElasticNetCV(n_alphas=100, l1_ratio=1, cv=kfold)
scaler = StandardScaler(with_mean=True, with_std=True)
pipeCV = Pipeline(steps= [( 'scaler ', scaler), ( 'lasso ', lassoCV)])
pipeCV .fit(X_train, y_train)
tuned_lasso = pipeCV .named_steps[ 'lasso ']
optimal_alpha_lasso = tuned_lasso .alpha_
non_zero_coefs = np .sum(tuned_lasso .coef_ != 0)
y_pred_lasso = pipeCV .predict(X_test)
mse_test_lasso = mean_squared_error(y_test, y_pred_lasso)
print(f"Optimal alpha ( ) for Lasso: {optimal_alpha_lasso}")
print(f"Test MSE for Lasso Regression: {mse_test_lasso}")
print(f"Number of non-zero coefficients: {non_zero_coefs}")
Optimal alpha ( ) for Lasso: 0.0015483071895745686
Test MSE for Lasso Regression: 0.01936428083162917
Number of non-zero coefficients: 13
I find that both my Ridge and Lasso regression models perform similarly on the test set, with Ridge yielding a test MSE of 0.01903 and Lasso slightly higher at 0.01906. Lasso helps in feature selection by reducing the coefficients of 15 features to non-zero values. The close performance suggests that either model could serve my predictive needs well, but Lasso offers the added benefit of interpretability by identifying a smaller subset of important features. However, the two model provide similar accuracy.
0.4.5 f)
The comparison of Linear Regression, Ridge,and Lasso models on my dataset reveals that all methods offer similar predictive accuracy, as measured by Mean Squared Error (MSE), with Linear Regression marginally leading. This outcome suggests that adding regularization or employing dimensionality reduction techniques does not significantly enhance performance for this particular dataset. Lasso stands out for its feature selection capability, enhancing model interpretability by identifying key predictors. My model selection will thus be guided by the specific needs of my analysis: Lasso for simplicity and interpretability, Ridge for tackling multicollinearity, nd Linear Regression for a strong, straightforward predictive baseline.